一, Dugangi ang pagkasensitibo sa sistema sa reaksyon:
1. Ilain ang taas nga kalidad nga RNA:
Ang malampuson nga synthesis sa cDNA naggikan sa taas nga kalidad nga RNA.Ang taas nga kalidad nga RNA kinahanglan labing menos bug-os nga gitas-on ug wala’y mga reverse transcriptase inhibitors sama sa EDTA o SDS.Ang kalidad sa RNA nagtino sa kinatas-ang gidaghanon sa sequence information nga imong ma-transcribe ngadto sa cDNA.Ang kasagarang paagi sa pagputli sa RNA mao ang usa ka lakang nga pamaagi gamit ang guanidine isothiocyanate/acid phenol.Aron malikayan ang kontaminasyon pinaagi sa pagsubay sa gidaghanon sa RNase, ang RNA nga nahimulag gikan sa mga sample nga dato sa RNase (sama sa pancreas) kinahanglan nga tipigan sa formaldehyde aron mapreserbar ang taas nga kalidad nga RNA, ilabi na alang sa dugay nga pagtipig.Ang RNA nga nakuha gikan sa atay sa ilaga sa batakan nadaot human gitipigan sa tubig sulod sa usa ka semana, samtang ang RNA nga nakuha gikan sa spleen sa ilaga nagpabilin nga lig-on human gitipigan sa tubig sulod sa 3 ka tuig.Dugang pa, ang mga transcript nga mas taas kaysa 4 kb mas sensitibo sa pagkadaot pinaagi sa pagsubay sa mga RNases kaysa gagmay nga mga transkrip.Aron madugangan ang kalig-on sa gitipigan nga mga sample sa RNA, ang RNA mahimong matunaw sa deionized formamide ug tipigan sa -70 ° C.Ang Formamide nga gigamit sa pagpreserbar sa RNA kinahanglan nga walay RNA-degrading debris.Ang RNA gikan sa pancreas mahimong mapreserbar sa formamide labing menos usa ka tuig.Kung nangandam sa paggamit sa RNA, mahimo nimong gamiton ang mosunud nga pamaagi aron ma-precipitate ang RNA: idugang ang NaCl sa 0.2M ug 4 ka beses ang gidaghanon sa ethanol, ibutang sa temperatura sa kwarto sa 3-5 minuto, ug centrifuge sa 10,000 × g sa 5 minuto.
2. Gamita ang RNaseH-inactive (RNaseH-) reverse transcriptase:
Ang mga inhibitor sa RNase kanunay nga gidugang sa mga reverse transcription reactions aron madugangan ang gitas-on ug abot sa cDNA synthesis.Kinahanglan nga idugang ang mga inhibitor sa RNase sa panahon sa reaksyon sa synthesis sa una nga strand sa presensya sa usa ka buffer ug usa ka ahente nga pagkunhod (sama sa DTT), tungod kay ang proseso sa wala pa ang synthesis sa cDNA nagdenature sa tigpugong, sa ingon nagpagawas sa gigapos nga RNase nga makapaubos sa RNA.Ang mga inhibitor sa protina RNase nagpugong lamang sa pagkadaut sa RNA pinaagi sa RNase A, B, C, ug ayaw pagpugong sa RNase sa panit, busa pag-amping nga dili ipaila ang RNase gikan sa imong mga tudlo bisan pa sa paggamit niini nga mga inhibitor.
Ang reverse transcriptase nag-catalyze sa pagkakabig sa RNA ngadto sa cDNA.Parehong M-MLV ug AMV adunay endogenous nga kalihokan sa RNaseH dugang sa ilang kaugalingon nga kalihokan sa polymerase.Ang kalihokan sa RNaseH ug kalihokan sa polymerase nakigkompetensya sa usag usa alang sa hybrid strand nga naporma tali sa RNA template ug sa DNA primer o cDNA extension strand, ug gipaubos ang RNA strand sa RNA:DNA complex.Ang template sa RNA nga gipaubos sa kalihokan sa RNaseH dili na magsilbi nga epektibo nga substrate alang sa synthesis sa cDNA, nga nagpamenos sa ani ug gitas-on sa synthesis sa cDNA.Busa, mapuslanon nga wagtangon o mapakunhod pag-ayo ang kalihokan sa RNaseH sa reverse transcriptase.
SuperScript Ⅱ reverse transcriptase, RNaseH- MMLV reverse transcriptase ug thermoScript reverse transcriptase, RNaseH- AMV, makakuha og mas daghang kantidad ug mas full-length nga cDNA kay sa MMLV ug AMV.Ang pagkasensitibo sa RT-PCR maapektuhan sa gidaghanon sa cDNA synthesis.Ang ThermoScript mas sensitibo kay sa AMV.Ang gidak-on sa mga produkto sa RT-PCR limitado sa abilidad sa reverse transcriptase sa pag-synthesize sa cDNA, ilabi na sa pag-clone sa mas dagkong mga cDNA.Kung itandi sa MMLV, ang SuperScripⅡ labi nga nagpataas sa abot sa taas nga mga produkto sa RT-PCR.Ang RNaseH-reverse transcriptase usab adunay dugang nga thermostability, mao nga ang reaksyon mahimo sa mga temperatura nga mas taas kaysa sa normal nga 37-42 °C.Ubos sa gisugyot nga kondisyon sa synthesis, gamita ang oligo(dT) primer ug 10 μCi sa [α-P]dCTP.Ang kinatibuk-ang abot sa unang strand gikalkulo gamit ang TCA precipitation method.Ang bug-os nga gitas-on nga cDNA gisusi gamit ang gidak-on nga mga banda nga gi-excised ug giihap sa usa ka alkaline agarose gel.
3. Ipataas ang incubation temperature para sa reverse transcription:
Ang mas taas nga temperatura sa paglumlum makatabang sa pag-abli sa sekundaryong istruktura sa RNA, nga nagdugang sa abot sa reaksyon.Alang sa kadaghanan sa mga template sa RNA, ang paglumlum sa RNA ug mga primer sa 65 ° C nga walay buffer o asin, nga gisundan sa paspas nga pagpabugnaw sa yelo magwagtang sa kadaghanan sa mga sekondaryang istruktura ug magtugot sa mga primer nga magbugkos.Bisan pa, ang pipila nga mga template adunay mga sekondaryang istruktura, bisan pagkahuman sa denaturation sa kainit.Ang pagpadako niining lisud nga mga templates mahimong ipahigayon gamit ang ThermoScript Reverse Transcriptase ug ibutang ang reverse transcription reaction sa mas taas nga temperatura aron mapalambo ang amplification.Ang mas taas nga temperatura sa paglumlum mahimo usab nga magpataas sa pagka-espesipiko, labi na kung ang mga primer nga piho nga gene (GSP) gigamit alang sa synthesis sa cDNA (tan-awa ang Kapitulo 3).Kung naggamit sa GSP, siguroha nga ang Tm sa mga primer parehas sa gipaabot nga temperatura sa paglumlum.Ayaw gamita ang oligo(dT) ug random nga mga primer nga labaw sa 60°C.Ang mga random nga primer nanginahanglan paglumlum sa 25°C sulod sa 10 ka minuto sa dili pa motaas sa 60°C.Dugang pa sa paggamit sa mas taas nga reverse transcription temperature, ang specificity mahimo usab nga mapauswag pinaagi sa direktang pagbalhin sa RNA/primer mix gikan sa 65°C denaturation temperature ngadto sa reverse transcription incubation temperature ug pagdugang ug pre-warmed 2× reaction mixture (cDNA hot-start synthesis) .Kini nga pamaagi makatabang sa pagpugong sa intermolecular base pagpares nga mahitabo sa ubos nga temperatura.Ang daghang pagbag-o sa temperatura nga gikinahanglan alang sa RT-PCR mahimong pasimplehon pinaagi sa paggamit sa usa ka thermal cycler.
Ang Tth thermostable polymerase naglihok isip DNA polymerase sa presensya sa Mg2+ ug isip RNA polymerase sa presensya sa Mn2+.Mahimo kining huptan nga init sa pinakataas nga temperatura nga 65°C.Bisan pa, ang presensya sa Mn2 + sa panahon sa PCR nagpamenos sa pagkamatinud-anon, nga naghimo sa Tth polymerase nga dili kaayo angay alang sa high-precision amplification, sama sa pag-clone sa cDNA.Dugang pa, ang Tth adunay ubos nga reverse transcription efficiency, nga makapamenos sa pagkasensitibo, ug, tungod kay ang reverse transcription ug PCR mahimo nga adunay usa ka enzyme, ang pagkontrol sa mga reaksyon nga walay reverse transcription dili magamit aron itandi ang mga produkto sa amplification sa cDNA nga adunay kontaminasyon sa genomic DNA.Ang mga produkto sa amplification gibulag.
4. Mga additives nga nagpasiugda sa reverse transcription:
Ang mga additives lakip ang glycerol ug DMSO gidugang sa first-strand synthesis reaction, nga makapakunhod sa kalig-on sa nucleic acid double-strand ug makatangtang sa secondary structure sa RNA.Hangtod sa 20% glycerol o 10% DMSO mahimong idugang nga dili makaapekto sa SuperScript II o MMLV nga kalihokan.Ang AMV mahimo usab nga motugot hangtod sa 20% nga glycerol nga wala’y pagkawala sa kalihokan.Aron mapadako ang pagkasensitibo sa RT-PCR sa SuperScriptⅡ reverse transcription reaction, ang 10% nga glycerol mahimong idugang ug i-incubate sa 45°C.Kung ang 1/10 sa reverse transcription reaction product idugang sa PCR, nan ang konsentrasyon sa glycerol sa amplification reaction mao ang 0.4%, nga dili igo sa pagpugong sa PCR.
5. RNaseH pagtambal:
Ang pagtambal sa mga reaksyon sa synthesis sa cDNA nga adunay RNaseH sa wala pa ang PCR makadugang sa pagkasensitibo.Alang sa pipila ka mga templates, gituohan nga ang RNA sa cDNA synthesis reaction nagpugong sa pagbugkos sa mga produkto sa amplification, diin ang RNaseH nga pagtambal makadugang sa pagkasensitibo.Kasagaran, gikinahanglan ang pagtambal sa RNaseH kung gipadako ang mas taas nga tibuuk nga mga template sa target sa cDNA, sama sa low-copy tuberous scherosis II.Alang niining lisud nga template, ang RNaseH nga pagtambal nagpalambo sa signal nga gihimo sa SuperScript II o AMV-synthesized cDNA.Alang sa kadaghanan nga mga reaksyon sa RT-PCR, opsyonal ang pagtambal sa RNaseH, tungod kay ang lakang sa denaturation sa PCR sa 95 ° C sa kasagaran nag-hydrolyze sa RNA sa RNA: DNA complex.
6. Pagpauswag sa Gamay nga Pamaagi sa Pagsusi sa RNA:
Ang RT-PCR labi nga mahagiton kung gamay ra nga kantidad sa RNA ang magamit.Ang Glycogen nga gidugang isip usa ka carrier sa panahon sa RNA isolation makatabang sa pagdugang sa abot sa gagmay nga mga sample.Ang RNase-free glycogen mahimong idugang dungan sa pagdugang sa Trizol.Ang glycogen matunaw sa tubig ug mahimong tipigan sa tubig nga bahin nga adunay RNA aron matabangan ang sunod nga pag-ulan.Para sa mga sample nga ubos sa 50 mg sa tissue o 106 ka kultura nga mga selula, ang girekomendar nga konsentrasyon sa RNase-free glycogen mao ang 250 μg/ml.
Ang pagdugang sa acetylated BSA sa reverse transcription reaction gamit ang SuperScript II makadugang sa pagkasensitibo, ug alang sa gamay nga kantidad sa RNA, ang pagkunhod sa gidaghanon sa SuperScript II ug ang pagdugang sa 40 ka yunit sa RNaseOut nuclease inhibitor makadugang sa lebel sa detection.Kung gigamit ang glycogen sa proseso sa pag-inusara sa RNA, girekomenda gihapon nga idugang ang BSA o RNase inhibitor kung gamiton ang SuperScript II alang sa reverse transcription nga reaksyon.
二, Dugangi ang espesipiko sa RT-PCR
1. CND Asynthesis:
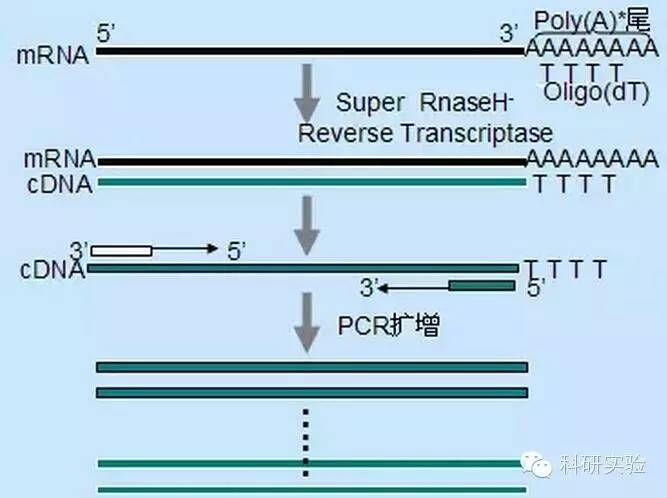
Ang first-strand nga cDNA synthesis mahimong masugdan gamit ang tulo ka lain-laing mga pamaagi, ang relatibong espesipiko nga makaapekto sa gidaghanon ug matang sa cDNA synthesized.
Ang random primer nga pamaagi mao ang pinakagamay nga espesipiko sa tulo ka mga pamaagi.Ang mga primer nag-anne sa daghang mga site sa tibuok transcript, nga nagmugna og mugbo, partial-length nga mga cDNA.Kini nga pamaagi kanunay nga gigamit aron makuha ang 5′ nga mga han-ay sa katapusan ug aron makuha ang cDNA gikan sa mga template sa RNA nga adunay mga rehiyon sa sekondaryang istruktura o adunay mga lugar sa pagtapos nga dili masundog sa reverse transcriptase.Aron makuha ang pinakataas nga cDNA, ang ratio sa mga primer sa RNA sa matag sample sa RNA kinahanglan nga matino sa empirikal nga paagi.Ang pagsugod nga konsentrasyon sa random primers gikan sa 50 ngadto sa 250 ng kada 20 μl nga reaksyon.Tungod kay ang cDNA nga gi-synthesize gikan sa kinatibuk-ang RNA gamit ang random nga mga primer mao ang panguna nga ribosomal RNA, ang poly(A) + RNA sa kasagaran gipili ingon template.
Ang mga primerong Oligo(dT) mas espesipiko kay sa mga random nga primer.Nag-hybrid kini sa poly(A) nga ikog nga makita sa 3′ nga tumoy sa kadaghanang eukaryotic mRNAs.Tungod kay ang poly(A)+ RNA gibana-bana nga 1% ngadto sa 2% sa kinatibuk-ang RNA, ang gidaghanon ug pagkakomplikado sa cDNA mas ubos kay sa random nga mga primer.Tungod sa taas nga espesipiko niini, ang oligo(dT) sa kasagaran wala magkinahanglan og pag-optimize sa ratio sa RNA sa mga primer ug poly(A)+ nga pagpili.Girekomenda nga gamiton ang 0.5μg oligo(dT) kada 20μl reaction system.Ang oligo(dT)12-18 angay alang sa kadaghanan sa RT-PCR.Ang ThermoScript RT-PCR System nagtanyag og oligo(dT)20 tungod sa mas maayo nga thermal stability niini para sa mas taas nga temperatura sa incubation.
Gene specific primers (GSP) mao ang pinakaspesipikong primer para sa reverse transcription step.Ang GSP usa ka antisense oligonucleotide nga espesipikong mag-hybrid sa RNA target sequence, dili sama sa random primers o oligo(dT), nga mo-anneal sa tanang RNAs.Ang parehas nga mga lagda nga gigamit sa pagdesinyo sa mga primer sa PCR magamit sa disenyo sa GSP sa mga reverse transcription nga reaksyon.Ang GSP mahimong parehas nga han-ay sa amplification primer nga nag-anne sa 3′-labing tumoy sa mRNA, o ang GSP mahimong gidisenyo aron ma-anneal sa ubos sa reverse amplification primer.Alang sa pipila nga gipadako nga mga hilisgutan, labaw pa sa usa ka antisense nga primer ang kinahanglan nga gidisenyo alang sa malampuson nga RT-PCR tungod kay ang ikaduha nga istruktura sa target nga RNA mahimong makapugong sa pagbugkos sa primer.Girekomenda nga gamiton ang 1 pmol antisense GSP sa usa ka 20 μl first-strand synthesis reaction.
2. Ipataas ang incubation temperature para sa reverse transcription:
Aron mapahimuslan sa hingpit ang bug-os nga bentaha sa espesipiko sa GSP, ang usa ka reverse transcriptase nga adunay mas taas nga thermostability kinahanglan gamiton.Ang mga thermostable nga reverse transcriptases mahimong ma-incubate sa mas taas nga temperatura aron madugangan ang kahigpit sa reaksyon.Pananglitan, kung ang usa ka GSP mag-anne sa 55°C, ang espesipiko sa GSP dili hingpit nga magamit kung ang AMV o M-MLV gigamit alang sa reverse transcription sa ubos nga stringency nga 37°C.Bisan pa, ang SuperScript II ug ThermoScript mahimong ma-react sa 50 ° C o mas taas, nga magwagtang sa dili piho nga mga produkto nga namugna sa mas ubos nga temperatura.Para sa pinakataas nga espesipiko, ang RNA/primer mix mahimong direktang ibalhin gikan sa 65°C denaturation temperature ngadto sa reverse transcription incubation temperature ug idugang sa pre-warmed 2× reaction mix (cDNA synthesis hot start).Kini makatabang sa pagpugong sa intermolecular base pagpares sa ubos nga temperatura.Ang daghang mga pagbag-o sa temperatura nga gikinahanglan alang sa RT-PCR mahimong pasimplehon pinaagi sa paggamit sa usa ka thermal cycler.
3. Mapakunhod ang kontaminasyon sa genomic DNA:
Ang posibleng kalisud nga masugatan sa RT-PCR mao ang kontaminasyon sa genomic DNA sa RNA.Ang paggamit sa maayo nga paagi sa pag-inusara sa RNA, sama sa Trizol Reagent, makapakunhod sa gidaghanon sa genomic DNA nga makahugaw sa pag-andam sa RNA.Aron malikayan ang mga produkto nga nakuha gikan sa genomic DNA, ang RNA mahimong matambalan gamit ang amplification-grade DNase I aron matangtang ang kontaminado nga DNA sa dili pa i-reverse transcription.Ang pagtunaw sa DNase I gitapos pinaagi sa paglumlom sa mga sample sa 2.0 mM EDTA sulod sa 10 minuto sa 65 ° C.Ang EDTA maka-chelate sa mga magnesium ions, nga makapugong sa magnesium ion-dependent nga RNA hydrolysis sa taas nga temperatura.
Aron mabulag ang gipadako nga cDNA gikan sa pagkontaminar sa genomic DNA amplification nga mga produkto, ang mga primer mahimo nga gidisenyo nga ang matag anneal sa pagbulag sa mga exon.Ang mga produkto sa PCR nga nakuha gikan sa cDNA mas mubu kaysa sa nakuha gikan sa kontaminado nga genomic DNA.Dugang pa, usa ka eksperimento sa pagkontrol nga walay reverse transcription ang gihimo sa matag RNA template aron mahibal-an kung ang usa ka gihatag nga tipik nakuha gikan sa genomic DNA o cDNA.Ang produkto sa PCR nga nakuha nga walay reverse transcription nakuha gikan sa genome.
Panahon sa pag-post: Mayo-16-2023








