Espesipiko sa detection
Sa kadaghanan nga mga kaso, ang katuyoan sa primer nga disenyo mao ang pagpadako sa pagkatino sa PCR.Gitino kini sa daghan o dili kaayo matag-an nga impluwensya sa daghang mga variable.Usa ka importante nga baryable mao ang han-ay sa 3′katapusan sa primer.
Importante, ang PCR assays nga gidisenyo alang sa specificity mas lagmit nga magpadayon sa taas nga episyente sa usa ka halapad nga dinamikong range, tungod kay ang assay dili makagama og dili piho nga mga produkto sa amplification, sa ingon nakigkompetensya sa PCR reagents o nagpugong sa nag-unang reaksyon sa amplification.
Siyempre, sa pipila ka mga kaso, ang espesipiko dili ang labing importante, pananglitan, kung ang tumong mao ang pag-ihap sa suod nga kalambigitan apan lain-laing mga pathogens, espesyal nga disenyo, optimization ug verification nga mga sumbanan gikinahanglan.
Ang melting curve kay usa ka standard nga pamaagi para sa pag-assess sa specificity sa mga amplicons, at least sa mga termino sa pagpadako sa usa ka target.Bisan pa, kinahanglan nga hatagan og gibug-aton nga ang mga kurba sa pagtunaw mahimong makapahisalaag tungod kay, pananglitan, mahimo silang maapektuhan sa hiniusa nga mga epekto sa suboptimal nga mga primer ug ubos nga konsentrasyon sa template.
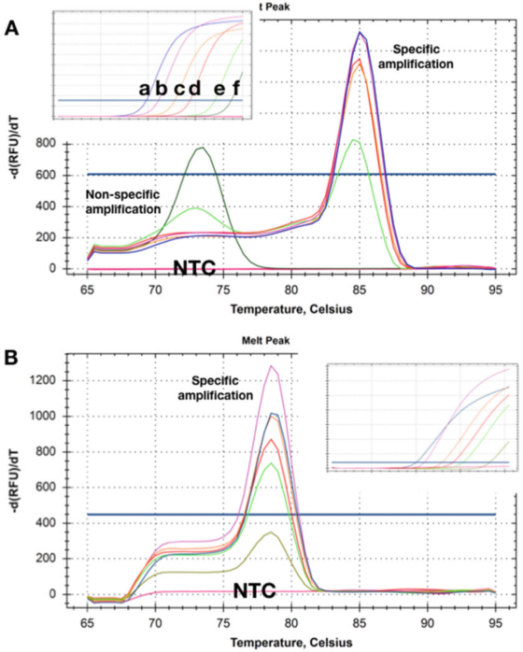
P5 |Ang melting curve nagpakita sa Tm shifts nga nakuha gikan sa duha ka detection sa lain-laing kantidad sa duha ka target nga DNA.
A. Sa mas taas nga konsentrasyon (ad)), walay dayag nga primer dimer human makompleto ang pagsukod sa qPCR.Samtang ang template nga konsentrasyon mikunhod ngadto sa 50 ka kopya (e), ang usa ka dili espesipiko nga produkto nagsugod sa pagpakita ug nahimong bugtong produkto sa pinakaubos nga konsentrasyon (f).
B. Ang pagsulay nagtala sa samang Tms sa tanang target nga konsentrasyon, ug walay dayag nga primer dimer bisan sa kinaubsang konsentrasyon (5 ka kopya).Kung gigamit kining duha ka pamaagi sa pag-ila, walay mga produkto sa amplification nga nakita sa mga NTC.
Ang P5 nagpakita sa mga dissolution curves nga nakuha sa mga sample diin ang template anaa sa lain-laing mga konsentrasyon.Gipakita sa P 5a nga sa duha ka labing ubos nga konsentrasyon, ang Tms sa dili piho nga mga produkto sa pagpadako nga gihimo mas ubos kaysa sa piho nga mga amplicon.
Dayag, kini nga paagi sa pag-ila dili kasaligan nga magamit aron makit-an ang mga target nga anaa sa ubos nga konsentrasyon.
Makapainteres, ang mga NTC, ie, mga sample nga wala’y DNA, wala magrekord (dili piho) nga mga produkto sa amplification, nga nagpakita nga ang background nga genomic DNA mahimong makaapil sa dili piho nga amplification / polimerisasyon.
Usahay ang ingon nga mga primera sa background ug dili piho nga pagpadako dili masulbad, apan kanunay nga posible ang pagdesinyo sa usa ka pamaagi sa pag-ila nga wala’y dili piho nga pagpadako sa bisan unsang konsentrasyon sa template ug NTC (P 5b).
Dinhi, bisan ang pagrekord sa pagpadako sa target nga konsentrasyon nga adunay usa ka Cq nga 35 maghimo usa ka piho nga kurba sa dissolution.Sa susama, ang mga NTC walay gipakita nga mga timailhan sa dili piho nga pagpadako.Usahay, ang pamatasan sa pag-ila mahimong magdepende sa inahan nga ilimnon, ug ang dili piho nga pagpadako lamang ang nakit-an sa pipila nga mga komposisyon sa buffer, nga mahimo’g adunay kalabotan sa lainlaing mga konsentrasyon sa Mg2+.
Kalig-on sa detection
Ang pag-optimize sa Ta usa ka mapuslanon nga lakang sa empirical nga pag-verify ug proseso sa pag-optimize sa qPCR detection.Naghatag kini usa ka direkta nga timailhan sa kalig-on sa primer set pinaagi sa pagpakita sa temperatura (o temperatura range) nga nagpatungha sa labing ubos nga Cq nga wala gipadako ang NTC.
Ang duha ngadto sa upat ka pilo nga kalainan sa pagkasensitibo mahimong dili importante alang sa mga tawo nga adunay taas nga mRNA nga ekspresyon, apan alang sa diagnostic nga mga pagsulay, kini mahimong magpasabot sa kalainan tali sa positibo ug sayop nga negatibo nga mga resulta.
Ang mga kabtangan sa Ta sa mga primer sa qPCR mahimong magkalainlain kaayo.Ang pipila ka mga pagsulay dili kaayo lig-on, ug kung dili kini himuon sa ilawom sa labing maayo nga kantidad sa Ta sa mga primer, dali silang mahugno.
Importante kini tungod kay kini nga matang sa detection kasagaran adunay problema sa tinuod nga kalibutan, ug ang kaputli sa sample, ang konsentrasyon sa DNA, o ang presensya sa ubang DNA mahimong dili maayo.
Dugang pa, ang gipunting nga numero sa kopya mahimong magkalainlain sa usa ka halapad nga sakup, ug ang mga reagents, plastik nga mga galamiton, o mga instrumento mahimong lahi sa gigamit sa pag-set up sa pagsulay.
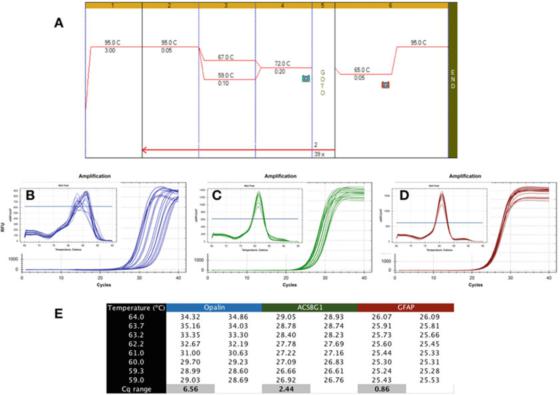
P6|Ang temperatura gradient nagpakita sa lain-laing kalig-on sa PCR detection.
A. Gamita ang Bioline's Sensifast SYBR mastermix (catalog number BIO-98050) sa paghimo sa PCR sa cDNA nga giandam gikan sa utok sa tawo nga RNA.
B. Gamita ang instrumento sa CFX qPCR sa Bio-Rad para irekord ang amplification map ug dissolution curve sa apalene (NM_033207, F: GCCATGGAGGAAAGTGACAGACC, R: CTCATGTGTGGGTGATCTCCTAGG).
C. Amplification graph ug melting curve sa ACSBG1 (NM_015162.4, F: CTACACTTCCGGCACCACTGG, R: GTCCACGTGATATTGTCTTGACTCAG).
D. Amplification graph ug dissolution curve sa GFAP (NM_002055.5, F: TGGAGAGGAAGATTGAGTCGCTGG, R: CGAACCTCCTCCTCGTGGATCTTC).
E. Cqs natala sa lain-laing annealing temperatura, nga nagpakita sa kalainan sa Cq natala ubos sa usa ka 7C temperatura gradient.
Ang P 6 nagpakita sa usa ka tipikal nga resulta sa usa ka dili gusto nga pagsulay, diin ang qPCR gihimo gamit ang usa ka gradient Tas tali sa 59C ug 67C (P 6a), gamit ang mga primer para sa tulo ka mga gene nga espesipiko sa utok sa tawo.
Makita gikan sa amplification graph nga ang mga primera sa Opalin layo sa ideal tungod kay ang ilang labing maayo nga range sa Ta kay hiktin kaayo (Figure 6b), nga mao, ang mga Cq kay kaylap nga nagkatibulaag, nga miresulta sa Cqs nga kamahinungdanon itandi sa ilang labing maayo nga Cqs Low.
Kini nga pamaagi sa pag-ila dili lig-on ug mahimong mosangpot sa suboptimal nga pagpadako.Busa, kini nga parisan sa mga primer kinahanglan nga bag-ohon ang disenyo.Dugang pa, ang pag-analisa sa kurba sa pagtunaw (inset) nagpakita nga ang espesipiko niini nga pamaagi sa pag-ila mahimo usab nga problema, tungod kay ang pagtunaw nga kurba sa matag Ta lahi.
Ang ACSBG1 detection method nga gipakita sa P 6c mas lig-on kay sa Opalin detection method sa ibabaw, apan layo pa kini sa ideal, ug lagmit nga kini mapauswag.
Bisan pa, gipasiugda namon nga wala’y kinahanglan nga koneksyon tali sa kalig-on ug espesipiko, tungod kay ang curve sa pagtunaw nga gihimo sa kini nga pamaagi sa pag-ila nagpakita sa parehas nga kantidad sa peak sa tanan nga Tas (inset).
Sa laing bahin, ang eksaminasyon sa kalig-on mas maagwanta, nga nagpatunghag susamang mga Cq sa usa ka halapad nga han-ay sa Tas, sama sa pagsulay sa GFAP nga gipakita sa P 6d.
Ang kalainan sa Cqs nga nakuha sa parehas nga 8 degree Celsius nga sakup dili mubu sa 1, ug ang dissolution curve (inset) nagpamatuod sa mga kinaiya sa pagkakita sa kini nga sakup sa temperatura.Angay nga matikdan nga ang kalkulado nga Tas ug ang aktuwal nga Ta range mahimong lahi kaayo.
Adunay daghang mga giya nga gidisenyo aron matabangan ang mga tigdukiduki sa pagdesinyo sa episyente nga mga primer, kadaghanan niini gibase sa dugay nang natukod nga mga lagda ug daghang pagtagad ang gihatag sa 3′end sa mga primer.Kanunay nga girekomenda nga ilakip ang usa ka G o C sa 3' katapusan ug duha ka G o C nga mga base (GC clamp), apan dili molapas sa duha sa katapusang 5 nga mga base.
Sa praktis, kini nga mga lagda makagiya sa mga tigdukiduki, apan kini dili kinahanglan nga husto ubos sa tanan nga mga kahimtang.
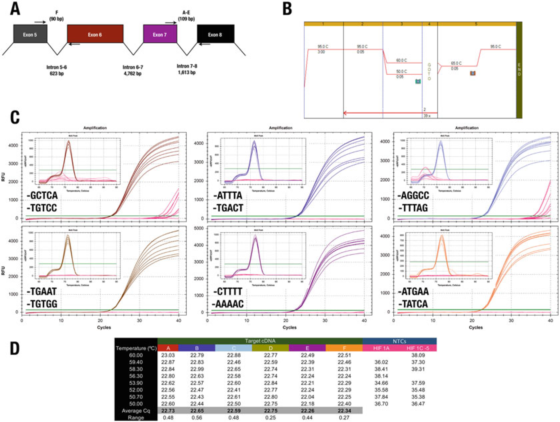
P7 |Ang 3′end sa primer adunay gamay nga epekto sa espesipiko o kahusayan.
A. Ang posisyon sa mga primer para sa gene sa HIF-1α (NM_181054.2) sa tawo.
B. Gamita ang Agilent Brilliant III SYBR Green mother liquor (Cat. No. 600882) aron madugangan ang unom ka test items.
C. Amplification graph ug melting curve nga natala sa Bio-Rad's CFX qPCR instrument ug 3′end primers.Ang mga NTC gipakita nga pula.
D. Cqs record sa matag test item
Pananglitan, ang resulta sa P 7 sukwahi sa 3′end nga lagda.Ang tanan nga mga disenyo nagmugna sa batakang parehas nga mga resulta, nga adunay duha lamang ka primerong kombinasyon nga motultol sa dili piho nga pagpadako sa NTC.
Bisan pa, dili namon masuportahan ang epekto sa clip sa GC, tungod kay sa kini nga kaso, ang paggamit sa A o T ingon usa ka maximum nga 30 nga mga base dili makapakunhod sa espesipiko.
Pagsulay C, diin ang F primer natapos sa GGCC, nagrekord sa Cqs sa NTCs, nagpakita nga ang usa ka tawo tingali gusto sa paglikay niini nga mga han-ay sa 30-katapusan.Gihatagan namon og gibug-aton nga ang bugtong paagi aron mahibal-an ang labing kaayo nga han-ay sa 3′end sa usa ka pares sa primer mao ang pagtimbang-timbang sa pipila nga mga primer sa kandidato nga eksperimento.
Episyente sa amplification
Mahinungdanon, bisan kung ang dili piho nga pagkakita sa PCR dili gyud mahimong espesipiko, ang kahusayan sa pagpadako mahimong mabag-o ug mapadako sa daghang lainlaing mga paagi pinaagi sa pagbag-o sa enzyme, ilimnon sa inahan, mga additives, ug mga kondisyon sa pagbisikleta.
Aron sa pagtimbang-timbang sa kaepektibo sa PCR detection, kini mao ang labing maayo sa paggamit sa usa ka serial dilution sa 10 o 5 ka beses sa target nucleic acid, nga mao, ang "standard curve pamaagi".
Kung gigamit ang mga PCR amplicon o sintetikong DNA nga target aron makamugna og standard curve, ang mga serial dilution niini nga mga target kinahanglang isagol sa kanunay nga gidaghanon sa background DNA (sama sa genomic DNA).
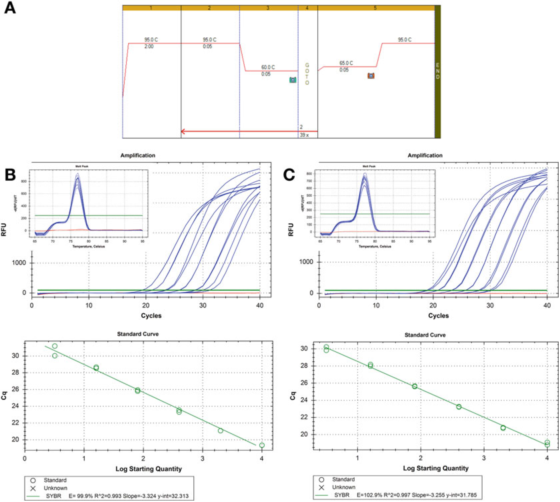
P8 |Dilution curve aron sa pagtimbang-timbang sa kahusayan sa PCR.
A. Gamita ang mga primer para sa HIF-1: F: AAGAACTTTTAGGCCGCTCA ug R: TGTCCTGTGGTGACTTGTCC ug Agilent's Brilliant III SYBR Green mastermix (catalog number 600882) para sa PCR ug melting curve nga mga kondisyon.
B. 100 ng RNA kay gibaliktad transcribe, lasaw 2 ka beses, ug serially lasaw cDNA samples lasaw 5 ka beses ngadto sa 1 ng tawo genomic DNA.Ang curve sa pagtunaw gipakita sa inset.
C. Ang reaksyon sa RT, dilution, ug serial dilution gisubli alang sa ikaduha nga sample sa cDNA, ug ang mga resulta parehas.
Ang P 8 nagpakita sa duha ka standard nga mga kurba, gamit ang parehas nga pamaagi sa pagkakita sa duha ka lain-laing mga sampol sa cDNA, ang resulta mao ang sama nga kahusayan, mga 100%, ug ang R2 nga bili susama usab, nga mao, ang ang-ang sa angay tali sa eksperimento nga datos ug ang linya sa regression o ang data Degree sa linearity.
Ang duha ka sumbanan nga mga kurba ikatandi, apan dili parehas.Kung ang katuyoan mao ang tukma nga pag-ihap sa target, kinahanglan nga matikdan nga dili madawat ang paghatag usa ka pagkalkula sa numero sa kopya nga wala ipasabut ang kawalay kasiguruhan.
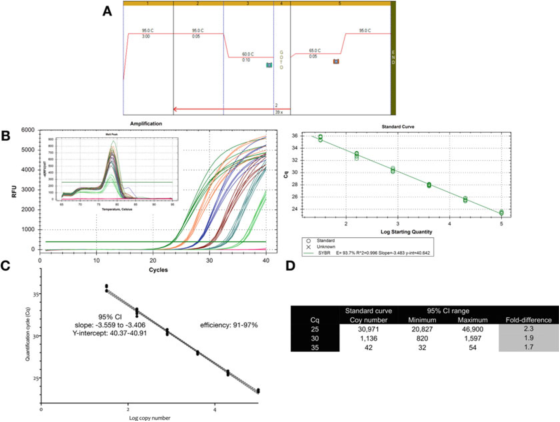
P9 |Ang kawalay kasiguruhan sa pagsukod nga may kalabotan sa pag-ihap gamit ang usa ka sukaranan nga kurba.
A. Gamita ang mga primer para sa GAPDH (NM_002046) sa paghimo sa PCR ug mga kondisyon sa pagtunaw sa kurba.F: ACAGTTGCCATGTAGACC ug R: TAACTGGTTGAGCACAGG ug Bioline's Sensifast SYBR mastermix (catalog number BIO-98050).
B. Amplification chart, melting curve ug standard curve nga natala gamit ang Bio-Rad's CFX qPCR instrument.
C. Standard curve graph ug 95% confidence interval (CI).
D. Ang numero sa kopya ug 95% nga agwat sa pagsalig sa tulo ka mga kantidad sa Cq nga nakuha gikan sa dilution curve.
Gipakita sa P 9 nga alang sa usa ka optimized nga pagsulay, ang kinaiyanhon nga kabag-ohan sa usa ka standard nga kurba gibana-bana nga 2 ka beses (95% nga agwat sa pagsalig, minimum hangtod sa labing taas), nga mahimo’g ang pinakagamay nga kabag-ohan nga mapaabut.
May kalabotan nga produkto:
Oras sa pag-post: Sep-30-2021